CGmapTools: DNA甲基化数据分析和可视化工具
Date: 2017年9月25日
By: 朱平
DNA甲基化与亚硫酸盐测序
在DNA甲基化转移酶(DNMT)的作用下,哺乳动物基因组上的胞嘧啶(C)第5位碳上会被加上一个甲基($-CH_3$),我们称之为DNA甲基化,简称5mC。作为表观遗传学的重要组成之一,DNA甲基化在许多生物过程中都扮演着非常重要的角色,如抑制转座子的活性、调控基因的表达、维持基因组印记、参与X染色体的失活等。
目前,亚硫酸盐转化结合高通量测序(Bisulfite conversion + High throughput sequencing)是检测DNA甲基化信号的金标准。其主要原理是:亚硫酸盐能特异性的将DNA上没有甲基化的C转化成U,通过PCR扩增我们最终检测到的信号为T,但是有甲基化的C则不会被改变。因此,通过比较测序数据中C和T的比例,我们可以评估DNA甲基化的程度。

随着测序成本的降低以及新技术方法的开发,DNA甲基化的研究越来越多,如何高效存储、共享、分析和可视化DNA甲基化数据成为我们亟需解决的问题。此前,已有一些针对DNA甲基化数据分析的工具被开发出来,涵盖质量控制、比对、找差异甲基化区域、预测SNP位点、Allele特异的甲基化等。尽管组合现有工具能够解决我们大部分的分析需求,但由于不同工具没有统一的输入/输出格式,给整合分析造成了较大困难,且部分工具的性能还有待较大提升。
CGmapTools显著提升BS-seq数据中计算杂合SNV的精准度,支持链特异DNA甲基化等40种分析及可视化
郭伟龙博士(中国农业大学)和朱平博士(天津血研所/北京大学)等人合作开发了DNA甲基化数据分析工具包:CGmapTools,包含40个独立的命令行工具,提供了全面的DNA甲基化数据分析和可视化功能。目前该工作已经在生物信息学著名期刊Bioinformatics上以original paper形式发表。
Weilong Guo #, Ping Zhu , et al. (2017), CGmapTools improves the precision of heterozygous SNV calls and supports allele-specific methylation detection and visualization in bisulfite-sequencing data, Bioinformatics, btx595.
CGmapTools的特色
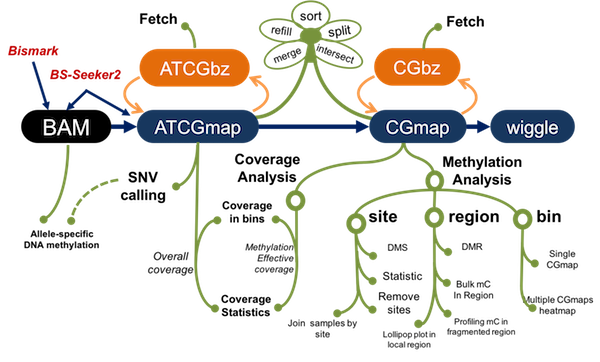
1. 统一的数据格式
该工作提出将ATCGmap和CGmap文件格式作为存储DNA甲基化信息的标准格式。CGmapTools提供的大部分高级分析都基于这两类文件进行。统一的文件格式接口将打破数据格式的屏障,有助于DNA甲基化组学领域实现数据共享和互通。
目前,CGmapTools支持对BS-Seeker2生成的CGmap和ATCGmap文件直接分析;也支持将Bismark的比对结果(BAM文件)进行解读为CGmap和ATCGmap格式的文件,或对Bismark的甲基化信息格式转化为CGmap格式。
示例:
|
|
2. 命令行模式轻松支持功能扩展
支持标准输入(STDIN)和输出(STDOUT),轻松实现工具的多重组合。
示例:
|
|
3. 支持快速检索
在ATCGmap和CGmap文件基础之上,我开发了对应的二进制压缩文件格式ATCGbz和CGbz,可以快速检索指定基因组区域的DNA甲基化状态,同时在保留主要DNA甲基化组信息的同时,大大减少了硬盘存储的占用空间(例如,对于WGBS来说,BAM文件需50G,CGmap压缩格式需5.6G,CGbz格式只需3.9G)。
此功能受到了
samtools view功能的启发。
示例:
|
|
4. 优化差异甲基化区域分析
针对基因组覆盖度较低的全基因组甲基化测序数据(WGBS)和覆盖不连续的简化甲基化测序数据(RRBS),该工作提出了动态片段化(dynamic fragment)的策略,实现数据的有效比较。
示例:
|
|
5. BS-Seq数据准确计算SNV新方法
利用ATCGmap文件的信息,通过引入wildcard的基因型,结合贝叶斯模型和二项分布模型设计了BayesWC和BinomWC两种SNV calling策略,使得从DNA甲基化数据计算heterozygous SNV的precision从之前80%提高到99%。
示例:
|
|
6. 支持Allele特异的甲基化分析和可视化
利用高精确度的杂合SNV作为输入,分析Allele-specific DNA methylation (ASM),并通过Tanghulu(“糖葫芦”)图对ASM区域的读段上DNA甲基化的状态进行直观展示。
示例:
|
|
7. 提供多层次甲基化分析和可视化工具
支持基于不同水平的DNA甲基化分析和可视化,如不同C的类型(CpG、非CpG等)、不同基因组区域(如基因区域、启动子区域等)、不同窗口大小、不同样本等。
|
|
##CGmapTools的安装
从CGmapTools的github主页上下载最新版本安装包,解压缩并安装。
|
|
CGmapTools充分利用了DNA甲基化数据的特性(即同时包含甲基化和基因组序列信息),从多组学角度提供了针对不同生物问题的分析功能。以下介绍CGmapTools在不同生物问题分析中的应用示例。
更多使用方法,可参考CGmapTools的网上教程。
